Biología - 16 - Genética Molecular
Concepto Molecular de Gen
La información hereditaria es aquella que pasa de una célula a su descendencia por división (mitosis, meiosis, gemación, bipartición, pluripartición...) o por fecundación.
Al expresarse, la información hereditaria...
- permite la autoconstrucción de cada célula y de cada ser vivo;
- permite el correcto funcionamiento de cada célula y de cada ser vivo;
- determina, conjuntamente con los factores del entorno, las características fenotípicas del individuo.
El material hereditario son las moléculas que contienen la información genética. En todos los seres vivos el material hereditario son moléculas de ADN. Esto es así porque el ADN...
- es capaz de replicarse, lo que le permite pasar la información genética a la descendencia celular;
- es una molécula químicamente estable, que conserva la información genética con gran fidelidad durante toda la vida de cada individuo;
- pese a lo anterior, es también una molécula que puede experimentar cambios (mutaciones), que es lo que ha permitido a los seres vivos adaptarse a los cambios en el entorno, y generar así la gran diversidad de especies y de individuos que conocemos.
En los eucariontes y las arqueas las moléculas de ADN están asociadas a unas proteínas, las histonas, formando un complejo macromolecular que se llama cromatina. La cromatina está en el núcleo de las células eucarióticas y en el citoplasma de las procarióticas. Cuando una célula se va a dividir, la cromatina sufre un proceso de plegamiento y compactación que da como resultado la formación de cromosomas.
En términos moleculares, un gen es un sector de una cadena de ADN que contiene, codificada en su secuencia de nucleótidos, información para la síntesis de alguna de las siguientes moléculas:
- una molécula de ARN mensajero;
- una molécula de ARN ribosómico;
- una molécula de ARN de transferencia.
La misión de los 3 tipos de ARN es colaborar en la síntesis de polipéptidos:
- cada molécula de ARNm determina la secuencia de aminoácidos de un polipéptido;
- el ARNr forma parte del ribosoma, que es donde se fabrica el polipéptido a partir de aminoácidos libres;
- el ARNt lleva los aminoácidos hasta el ribosoma y los coloca en el lugar exacto de la cadena polipeptídica.
Posteriormente cada polipéptido pasará por una serie de modificaciones postraduccionales en el REr y en el Aparato de Golgi, que lo convertirán en una proteína funcional.
Por eso se dice que los genes codifican para proteínas, aunque en realidad estos son solo los genes que codifican directamente para la síntesis de ARNm, y que se denominan genes codificantes.; estos son unos 20.400 en humanos, y suponen un 2% del total de nuestro genoma. Pero además hay:
- genes no codificantes: los que codifican para la síntesis de ARNr y ARNt;
- genes reguladores, que determinan cúando y en qué células se ha de expresar un gen codificante;
- transposones o "elementos saltadores", posibles precursores de los virus;
- pseudogenes: secuencias parciales o completas de genes que han quedado inactivados a lo largo de la evolución al acumular mutaciones;
- ADN repetitivo, de función estructural, como el de los telómeros y los centrómeros de los cromosomas.
A los 3 últimos elementos se les llama ADN silencioso, por carecer de papel bioquímico, o no ser conocido.
En los eucariontes y las arqueas los genes contienen sectores no codificantes, los intrones, intercalados entre las secuencias codificantes, los exones. Los intrones han de ser eliminados durante el proceso de maduración del ARNm antes de que este pueda ser leido en un ribosoma. Pero, lejos de ser inútiles, permiten la recombinación por sobrecruzamiento durante la profase I de la meiosis entre segmentos de un mismo gen. Como cada exón codifica para una región diferente de la proteína, se pueden generar así muchas variantes algo diferentes de esa misma proteína. Esto genera mucha diversidad fenotípica: es el mecanismo que permite que seamos capaces de producir más de 1 000 000 de anticuerpos diferentes. Por ello, la presencia de intrones favorece la variabilidad genética y la adaptación al entorno y promueve la evolución biológica.
| Bacterias | Arqueas | Eucariontes | |
|---|---|---|---|
| ADN dentro de compartimentos membranosos (núcleo, mitocondrias, cloroplastos) | No | No | Sí |
| ADN silencioso | Apenas | Apenas | Mucho |
| ADN asociado a histonas | No | Sí | Sí |
| Intrones en los genes | No | Sí | Sí |
La Replicación del ADN
Consiste en la biosíntesis de una copia de una molécula de ADN bicatenario idéntica a la original.
Transcurre en los lugares de la célula donde está el ADN, que en las células eucarióticas es mayoritariamente el núcleo. Tiene lugar durante la fase S de la interfase del ciclo celular. Es un acontecimiento previo a la división celular, que permite que cada una de las células hijas que se van a formar por mitosis reciba la misma información genética que tenía la célula madre de la que proceden.
Vamos a estudiar cómo sucede en las bacterias y después las particularidades de los eucariontes.
Etapas de la Replicación en las Bacterias
Inicio
El proceso comienza con el desapareamiento de las dos hebras de la molécula de ADN bicatenario, que comienza en un lugar concreto del cromosoma llamado origen de replicación. El desapareamiento es llevado a cabo por una enzima llamada helicasa, que, de modo análogo a como se abre una cremallera, va rompiendo los enlaces por puente de hidrógeno entre las bases complementarias de las hebras del ADN, que van quedando así separadas.
La región de la molécula de ADN que va quedando desapareada por acción de la helicasa se llama burbuja u ojo de replicación. La burbuja de replicación va expandiéndose por el cromosoma en ambos sentidos hasta que sus extremos contactan entre sí. Cada uno de los dos extremos de la burbuja de replicación, allí donde una helicasa va rompiendo los enlaces por puente de hidrógeno entre las bases, se denomina horquilla de replicación.
La región que queda desapareada queda también desenrollada, pero a costa de originar superenrollamientos en el resto de la molécula (como en una goma del pelo), los cuales son deshechos por otras enzimas, las topoisomerasas.
Para evitar que las bases desapareadas por la helicasa vuelvan a asociarse inmediatamente, hay unas proteínas, llamadas proteínas estabilizadoras o proteínas SSB, que se asocian con las regiones desapareadas del ADN en la burbuja de replicación.
Síntesis del ADN de las hebras hijas
Las zonas desapareadas de la molécula de ADN parental van a ser el molde o patrón sobre el que se van a sintetizar dos nuevas hebras de ADN, llamadas hebras hijas. Se van a ir añadiendo nucleótidos complementarios uno a uno hasta que las dos hebras hijas se terminen de sintetizar, de tal modo que al final habrá dos moléculas hijas de ADN bicatenario, cada una de ellas formada por una hebra parental y por una hebra hija; es por esto que se dice que el mecanismo de replicación del ADN es semiconservativo.
La síntesis de cada nueva hebra la realizan enzimas con actividad ADN polimerasa. La más habitual e importante en bacterias es la ADN-polimerasa III. Esta actúa así:
- Necesita la presencia de iones Mg2+ libres en el medio para poder actuar.
- Toma dNTP (desoxirribonucleótidos trifosfato) libres en el medio.
- Los hidroliza a dNMP + 2 Pi al romper sus 2 enlaces éster-fosfato, lo que permite liberar la energía necesaria para el proceso.
- Añade el dNMP al extremo 3' de la hebra hija en crecimiento, por lo tanto cada hebra hija va creciendo en sentido 5'→3', y lo hace en función de la complementariedad de bases entre hebra patrón y hebra hija: enfrente de dAMP coloca dTMP; enfrente de dCMP coloca dGMP; etc.
- Fabrica el enlace fosfodiéster entre el nuevo nucleótido y el nucleótido del extremo 3' de la hebra hija.
Pero la ADN-polimerasa III no puede comenzar la síntesis de una nueva hebra de ADN sobre la hebra patrón; solo puede continuar la síntesis de una hebra polinucleotítica ya comenzada; a este pequeño polinucleótido inicial se le llama cebador o primer. En el proceso de la replicación del ADN los cebadores son siempre moléculas de ARN de unos 10 nucleótidos de longitud, son sintetizados por una ARN-polimerasa denominada primasa y posteriormente han de ser sustituidos por ADN.
Hebras conductoras y retardadas
La ADN-polimerasa III solo puede añadir dnucleótidos a una cadena de ADN en sentido 5'→3'. Esto plantea un problema, ya que en cada horquilla de replicación se sintetizan 2 hebras nuevas de ADN y, como es evidente, solo una de las dos, la llamada hebra conductora, puede crecer en sentido 5'→3'. La otra, que se llama hebra retardada, necesariamente ha de crecer en sentido 3'→5'.
Esta hebra no crece de forma continua, sino a trozos: estos se llaman fragmentos de Okazaki y tienen una longitud de 1000 a 2000 nucleótidos. Y son sintetizados por la ADN-polimerasa III en sentido 5'→3', es decir, en sentido contrario al de crecimiento de la propia hebra. Así pues, la hebra retardada crece del mismo modo que crece una cola de espera, al irse añadiendo gente por detrás.
Como ambas hebras hijas van creciendo en ambos sentidos al ir avanzando cada una de las dos horquillas de la burbuja de replicación, se dice que la replicación del ADN es bidireccional. Y por el mismo motivo, una misma hebra es sintetizada de forma conductora en una horquilla y de forma retardada en la otra.
Eliminación de los cebadores
Para la hebra conductora solo hace falta un único ARN cebador. En cambio en la hebra retardada hace falta un cebador para cada fragmento de Okazaki. Los cebadores, como se ha dicho, son moléculas cortas de ARN sintetizadas por la primasa. Cada cebador, una vez cumplida su función, es eliminado y sustituido por ADN por la enzima ADN-polimerasa I (que por lo tanto tiene tanto actividad exonucleasa como actividad polimerasa de ADN). Una última enzima, la ADN-ligasa, se encarga de empalmar los diversos fragmentos de ADN de la hebra retardada, fabricando enlaces fosfodiéster entre el nucleótido del extremo 3' del fragmento sintetizado por la ADN-polimerasa I y el nucleótido del extremo 5' del fragmento de Okazaki que tiene delante.
Finalización
El proceso termina cuando las dos horquillas del ojo de replicación establecen contacto entre sí. En ese momento se completa la síntesis de las hebras hijas. Tras ello se separan las dos moléculas hijas de ADN bicatenario, cada una de ellas formada por una hebra hija y por una hebra parental.
La Replicación en los Eucariontes (No EBAU)
La replicación en eucariontes es similar a la descrita para procariontes, con las siguientes diferencias:
- En el cromosoma bacteriano hay solo un origen de replicación y por lo tanto una sola burbuja de replicación. En los cromosomas eucarióticos hay muchos orígenes de replicación: más de 100 en cada cromosoma humano, cada uno de los cuales da lugar a un ojo de replicación. Todos los orígenes de replicación de cada cromosoma se activan simultáneamente. Esto permite alcanzar mayor velocidad en la replicación lo que resulta necesario al tener los eucariontes mucho más ADN que las bacterias.
- En procariontes las moléculas de ADN (y los cromosomas) son circulares. En eucariontes son lineales; por ello aquí la replicación termina cuando todos los ojos de replicación se han unido entre sí y los dos de los extremos alcanzan los extremos de la molécula.
- En bacterias durante la replicación el ADN está básicamente desnudo (sin asociarse a proteínas). En eucariontes durante la replicación el ADN está asociado a las histonas, las cuales entorpecen el proceso de replicación del ADN, haciendo que los fragmentos de Okazaki sean más pequeños y que la velocidad de replicación en cada horquilla también lo sea (unas 10 veces menor en ambos casos).
- Las histonas originales se quedan siempre en la hebra conductora; para la hebra retardada se sintetizan unas nuevas.
- En procariontes hay tres ADN polimerasas diferentes: I, II y III. En eucariontes hay cinco.
La Transcripción del ADN
Consiste en la biosíntesis de una molécula específica de ARN utilizando un gen como molde, lo que significa que la síntesis de cada molécula de ARN se realiza de forma que su secuencia de bases sea complementaria de la del ADN de un gen concreto.
Transcurre en los lugares de la célula donde está el ADN, que en las células eucarióticas es mayoritariamente el núcleo; desde aquí el ARN transcrito ha de migrar hasta el citoplasma para participar en la traducción.
Vamos a ver cómo ocurre la transcripción en las bacterias y después veremos las particularidades de los eucariontes.
Etapas de la Transcripción en las Bacterias
Iniciación
La ARN-polimerasa se asocia a una región del ADN denominada promotor, que no se va a transcribir, pero que está situada al lado del extremo 3' del gen que se va a transcribir. El promotor contiene unas secuencias de nucleótidos denominadas secuencias consenso, que son reconocidas por la ARN-polimerasa y que son similares para todos los genes de cada especie. A veces una región promotora es común a varios genes (hay una unidad de transcripción policistrónica), por lo que la ARN-polimerasa transcribirá todos ellos, uno detrás de otro.
La ARN-polimerasa desenrolla entre 1 y 2 vueltas de la doble hélice de ADN. De las dos hebras de la región de ADN que se va a transcribir, una se denomina hebra molde o patrón, y es la que contiene la secuencia promotora a la que se ha asociado la polimerasa, y la que va a servir como molde del ARN que va a ser sintetizado por la ARN-polimerasa, al ir esta añadiendo uno a uno los nucleótidos complementarios a los del gen. La otra se denomina hebra codificante o informativa, y es idéntica al ARN transcrito (salvo U por T y ribosa por desoxirribosa).
La ARN-polimerasa no necesita cebador por lo que añade directamente el primer nucleótido.
Alargamiento
La ARN-polimerasa se va desplazando a lo largo del gen en sentido 3→5', al tiempo que va fabricando la cadena de ARN en sentido 5→3': va añadiendo uno a uno los nucleótidos complementarios a los del gen, tras lo cual fabrica el enlace fosfodiéster entre el nuevo nucleótido y el nucleótido del extremo 3' de la cadena de ARN en crecimiento. Para ello la ARN-polimerasa va tomando NTP libres en el medio, que al ir siendo añadidos a la cadena de ARN en crecimiento son hidrolizados a NMP + 2 Pi, obteniéndose la energía necesaria para la síntesis del enlace fosfodiéster de la ruptura de los dos enlaces éster-fosfato de alta energía de cada NTP.
La cadena de ARN va creciendo por su extremo 3' y permanece unida al ADN a lo largo de unos 12 pb; mientras tanto, por su extremo 5' el ARN se va separando de la cadena de ADN patrón. Al tiempo el ADN se va desespiralizando allí donde va llegando la polimerasa y reespiralizándose por la parte que va quedando detrás.
Terminación
Todos los genes en su extremo 5' tienen una secuencia de terminación (TTATTT, en genes eucarióticos) que es reconocida por la ARN-polimerasa y que determina que esta se separe de la cadena de ADN patrón. A partir de entonces ya no se añaden más nucleótidos a la cadena de ARN en crecimiento, y esta también se separa completamente de la cadena de ADN patrón.
La Transcripción en los Eucariontes (No EBAU)
- En las células eucarióticas la transcripción ocurre con el ADN asociado a las histonas (estado de fibra nucleosómica), salvo cuando se trata de genes que se transcriben muy frecuentemente.
- Los ARNm son siempre monocistrónicos (corresponden a un solo gen), mientras que en bacterias suelen ser policistrónicos.
- El ARN nucleolar se transcribe a partir de un gen repetido numerosas veces en tandem, situado en una regiones del ADN de algunos cromosomas denominadas en conjunto "ADN organizador nucleolar", ya que constituyen el nucleolo de esas células. Y ha de pasar por un proceso de maduración, que consiste en su fragmentación en los ARNr 28 S, 18 S y 5,8 S. Para formar los ribosomas todos ellos se ensamblarán con una serie de proteínas y con el ARNr 5 S, que ha sido sintetizado por un gen diferente, también repetido muchas veces.
- En las células procarióticas (y en las mitocondrias y los cloroplastos) solo hay una ARN-polimerasa. En el núcleo de las eucarióticas hay 3 ARN-polimerasas:
- ARN-polimerasa I (que fabrica el ARN nucleolar, precursor de la mayoría de los ARNr);
- ARN-polimerasa II (que sintetiza casi todo el ARNm); y
- ARN-polimerasa III (que sintetiza los ARNt, el ARNr 5S y el ARNm que será traducido a histonas).
- En eucariontes el ARN mensajero primario (pre-ARNm, ARN heterogéneo nuclear) ha de madurar antes de abandonar el núcleo por los poros de la envoltura nuclear. Esto incluye 3 procesos:
- Durante la fase de alargamiento, al ARNm se le añade en su extremo 5' una caperuza formada por el nucleótido 7-metil-GTP. Sirve para permiti el anclaje del ARNm al ribosoma para la traducción.
- Tras la fase de terminación, al extremo 3' de cada ARNm se le añade la cola de poli-A, una secuencia de unos 200 AMP que ayudan a evitar la degradación rápida del ARNm por las exonucleasas del citoplasma.
- Todos los ARNm (savo los de las histonas) contienen intrones, porque los contenían también los genes a partir de los cuales han sido transcritos. Y hay que eliminarlos antes de que el ARNm pueda ser traducido en los ribosomas. Esto se hace en un proceso llamado corte y empalme, que tiene 2 fases:
- una enzima llamada espliceosoma se une a los extremos de cada intrón, y lo extirpa del ARNm;
- una enzima ligasa empalma los exones.
La Biosíntesis de las Proteínas
La expresión del mensaje genético consiste en que la célula viva sintetice las proteínas que necesite en cada momento, gracias a la expresión de los genes correspondientes. Este proceso transcurre en dos etapas: la transcripción y la biosíntesis de proteínas. A su vez, la biosíntesis de proteínas tiene tres partes: (a) activación de los aminoácidos, (b) traducción o biosíntesis de polipéptidos y (c) modificaciones postraduccionales de los polipéptidos para dar lugar a una proteína activa.
Activación de los Aminoácidos
Para que sea posible la biosíntesis de los diversos polipéptidos, los aminoácidos que los constituyen han de ser transportados hasta los ribosomas.
Los encargados de este transporte son las diversas moléculas de ARN de transferencia, que se unen a los aminoácidos dando lugar a moléculas de aminoacil-ARNt.
- La asociación se produce entre el extremo C-terminal del aminoácido y el extremo 3' del ARNt.
- Esta unión es espécífica: cada clase de moléculas de ARNt se asocia a uno de los 20 tipos de aminoácidos que forman las proteínas de los seres vivos. Esta especificidad viene determinada por la secuencia de nucleótidos que cada molécula de ARNt lleve en su anticodón, que es un grupo de tres nucleótidos que hay en una determinada posición del bucle central de cada ARNt. Cada anticodón es complementario de un codón concreto de ARNm. Y así por ejemplo, al ARNt cuyo anticodón es 3'-GGC-5' le corresponde el aminoácido prolina y al ARNt cuyo anticodón es 3'-UAC-5' le corresponde la metionina.
- La reacción de asociación es catalizada por enzimas con actividad aminoacil-ARNt sintetasa. Hay una específica para cada aminoácido: por ejemplo: metionil-ARNt sintetasa, alanil-ARNt sintetasa, etc.
- Esta reacción consume energía (a causa del nuevo enlace químico que se forma), que es aportada por una molécula de ATP.
- La ecuación global es:
aa' + ARNt + ATP → aa'-ARNt + AMP + PPi
Traducción
La traducción es el proceso metabólico en el que, con el concurso de las tres clases de moléculas de ARN, se va a sintetizar en los ribosomas el polipéptido específico que corresponda a la secuencia de nucleótidos de un ARNm.
Como los ribosomas eucarióticos están en el citoplasma, separados del núcleo, en eucariontes la traducción sucede en un compartimento celular distinto de la transcripción, el citoplasma, y una vez que el ARNm haya madurado (eliminación de intrones y adición de caperuza y de cola de poli-A). En procariontes el ARNm no ha de madurar y la traducción puede comenzar incluso antes de que haya terminado la transcripción.
Todos los ARNm tienen una región central codificante, contenida entre dos regiones no codificantes, una en cada extremo de la molécula. Los ribosomas traducen la secuencia de nucleótidos de la región codificante del ARNm a una secuencia de aminoácidos de un polipéptido.
Esto ocurre del siguiente modo: el ribosoma "lee" la región codificante del ARNm en sentido 5'→3' y de tres en tres nucleótidos. A cada grupo de tres nucleótidos, leído en sentido 5'→3' se le denomina codón. Cada codón es, pues, como una "palabra" compuesta por tres "letras". Y el ribosoma va a ir añadiendo, uno a uno, los distintos aminoácidos que resulten de traducir cada uno de los codones del ARNm, según un código de correspondencias denominado código genético. Así por ejemplo, al codón 5'-CCU-3' le corresponde el aminoácido prolina y al codón 5'-AUG-3' le corresponde la metionina.
Los codones tienen 3 nucleótidos porque si tuviesen 2 solo habría 4x4 = 16 posibles combinaciones, insuficientes para codificar los 20 aminoácidos de las proteínas.
De las 64 combinaciones posibles de tres nucleótidos seguidos, 61 codones codifican para aminoácidos, pero 3 de ellos no lo hacen: UAA, UAG y UGA, que son los denominados tripletes sin sentido, y actúan como señales de terminación: sirven para indicarle al ribosoma donde concluye la región codificante de un ARNm para que no siga leyendo y deje de añadir aminoácidos.
Por su parte el codón AUG, además de codificar para el aminoácido metionina, sirve como señal de iniciación de la traducción: le indica al ribosoma donde comienza la región codificante de un ARNm.
Como tenemos 61 codones y solo 20 aminoácidos proteínicos, ha de haber codones a los que les corresponda el mismo aminoácido, esto es, hay codones que son sinónimos, por ejemplo, los codones CCA, CCU, CCC y CCG codifican para la prolina. Por eso se dice que el código genético es un código degenerado.
Además, el código genético, salvo muy raras excepciones (p.e. en mitocondrias y en micoplasmas) o variaciones (p.e. 5'-AUG-3' se traduce por formil-metionina en las bacterias), es universal, es decir, rige para todas las especies de seres vivos (y también para los virus). Así pues, en cualquier especie, 5'-CCU-3' siempre significa "prolina", y un mismo ARNm siempre será traducido a la misma proteína. Esto es lo que permite que, tras insertarle a una bacteria el gen (purificado de intrones) de la insulina humana, pueda expresarlo y fabricar insulina humana. Y es una prueba del origen común de todos los seres vivos.
Iniciación
Inicialmente, el ribosoma está disociado en sus dos subunidades. El ARNm que llega al ribosoma se asocia, por la secuencia líder de su extremo 5', al ARNr de la subunidad menor, y se desplaza a lo largo de ella hasta que el codón iniciador, que siempre es 5'-AUG-3', se ubique en el sitio P.
La subunidad ribosómica menor tiene 3 sitios significativos:
- Sitio A: lugar de llegada de los nuevos aminoacil-ARNt (salvo el 1º, que llega al sitio P).
- Sitio P: ubicación del ARNt que porta el péptido que se está sintetizando.
- Sitio E: lugar desde el que sale el penúltimo ARNt en llegar.
Al codón iniciador del ARNm se va a asociar mediante puentes de hidrógeno el anticodón de una molécula de metionil-ARNt, que es un aminoacil-ARN cuyo anticodón es 3'-UAC-5' y cuyo aminoácido es la metionina. De esto se deduce que el primer aminoácido de todos los polipéptidos recién sintetizados va a ser la metionina (o la formil-metionina). A este primer aminoácido se le llama aminoácido iniciador, y suele ser eliminado en la fase de modificación.
Por último a este grupo de moléculas se une la subunidad ribosómica mayor, con lo que se forma el complejo de iniciación.
Este proceso es catalizado por unas proteínas llamadas factores de iniciación, tiene un consumo de energía aportada por moléculas de GTP, y requiere la presencia de iones Mg2+.
Elongación
En esta fase se van a ir añadiendo uno a uno los aminoácidos que corresponden a todos los demás codones del ARNm. Para ello es necesario que el ribosoma y el ARNm se desplacen uno respecto del otro.
- El ribosoma va a avanzar hacia el extremo 3' del ARNm, moviéndose una distancia de 3 nucleótidos cada vez (translocación). Este paso no es necesario la primera vez, ya que al llegar el primer metionil-ARNt directamente al sitio P, el sitio A ya está libre.
- En cualquier otro caso, tras cada translocación el sitio A queda libre y a él llegará el aminoacil-ARNt complementario del codón que allí se encuentre.
- Tras ello, el anterior aminoácido en llegar (la metionina, si fue el primero), separa su grupo carboxilo de su ARNt y lo une mediante un enlace peptídico al grupo amino del que acaba de llegar al sitio A. Este proceso es catalizado por la enzima peptidil-transferasa. Ahora tenemos un peptidil-ARNt, que es la molécula compuesta por el péptido en crecimiento (que de momento solo es un dipéptido), unida por su extremo C-terminal a una única molécula de ARNt.
Después el ribosoma efectúa la traslocación correspondiente hacia el extremo 3' del ARNm, con lo que...
- el ARNt que portaba al anterior aminoácido en llegar (la metionina, si fue el primero) pasa al sitio E, desde donde se separa del complejo ribosomal.
- el peptidil-ARNt pasa de estar en el sitio A a estar en el sitio P;
- hay un nuevo codón en el sitio A, que queda libre, y al que habrá de llegar el siguiente aminoacil-ARNt;
Este proceso se repite una y otra vez al ir recorriendo el ribosoma el ARNm en sentido 5'→3'. En cada paso, el aminoacil-ARNt que entra al sitio A, añade a su aminoácido el péptido que se está formando y que se encuentra en el sitio P.
Este proceso es catalizado por una proteínas llamadas factores de elongación y tiene un consumo de energía aportada por moléculas de GTP.
Terminación
Cuando en el sitio A del ribosoma aparece el codón de terminación que haya al final de la región codificante del ARNm (UAA, UAG o UGA), se interrumpirá la elongación de la cadena polipeptídica, puesto que no hay ningún aminoacil-ARNt cuyo anticodón sea complementario de esos codones.
Sin embargo esos codones son reconocidos por los factores de liberación, que se asocian al sitio A y cuya acción provoca la separación del polipéptido que se acaba de sintetizar del ARNt al que estaba unido, y por lo tanto su liberación del ribosoma. Los restantes componentes del complejo ribosomal (ARNt del sitio P, ARNm y las dos subunidades ribosomales) también se separan unos de otros. Este proceso también consume energía, que es aportada por moléculas de GTP.
El primer aminoácido que se ha colocado (la metionina o la formil-metionina) es el que va a estar en el extremo N-terminal de la cadena polipeptídica, y el último en llegar va a estar en el extremo C-terminal.
Normalmente cada ARNm es traducido simultáneamente por varios ribosomas, uno detrás de otro. A este complejo formado por una molécula de ARNm y varios ribosomas, cada uno con su cadena polipeptídica en crecimiento, se le denomina polisoma o polirribosoma.
Modificaciones Postraduccionales
A medida que se va sintetizando una cadena polipeptídica, esta va adoptando una determinada conformación o estructura tridimensional al irse estableciendo los diversos enlaces internos entre los aminoácidos propios de las estructuras secundaria y terciaria de las proteínas (enlaces por puente de hidrógeno, enlaces iónicos, puentes disulfuro...). Estos plegamientos, que culminan dentro del REr, a veces ya dan como resultado una proteína activa. Pero con frecuencia son necesarias ciertas modificaciones adicionales dentro de las cisternas y sáculos del retículo endoplasmático rugoso y del aparato de Golgi:
- REr: en proteínas con estructura cuaternaria, como la hemoglobina, la asociación de varias cadenas polipeptídicas (protómeros) entre sí, para dar lugar a una proteína funcional.
- REr y Golgi: en heteroproteínas, como la hemoglobina, la asociación de la cadena polipeptídica a grupos prostéticos (glúcidos, lípidos, grupos fosfato, grupos hemo...) o cofactores (metales, coenzimas).
- Golgi: la eliminación de algún aminoácido o proteólisis (es muy frecuente la eliminación del aminoácido iniciador);
- Golgi: la modificación de la cadena lateral de algún aminoácido (por glicosilación, fosforilación, etc.);
La Regulación de la Expresión Génica (No EBAU)
¿Por qué es Necesaria?
La actividad de cada célula depende de la expresión de sus genes, es decir, de su transcripción y de la subsiguiente biosíntesis de proteínas. Pero todas las células somáticas de cada individuo tienen exactamente los mismos genes. Y mientras que unas células funcionan como células epiteliales, otras lo hacen como células nerviosas y así hasta completar los varios centenares de tipos celulares que tenemos.
La explicación está en que en cada tipo célular hay una importante cantidad de genes que no se expresan nunca; son genes que se localizan en la heterocromatina constitutiva: zonas de la cromatina especialmente condensadas e inaccesibles a las ARN-polimerasas. En cada célula solo se va a expresar el conjunto de genes que le permita funcionar como célula epitelial o como célula nerviosa, etc., que son aquellos que se localizan en las regiones más descondensadas de la cromatina.
Pero además, de entre todos los genes que se pueden expresar en cada célula, en unos momentos se expresarán unos y en otros momentos se expresarán otros, según convenga a las necesidades de la célula y a las del individuo al que esta pertenece. Los genes nunca se expresan de forma innecesaria, porque ello supone un gasto innecesario de recursos y porque puede tener efectos perjudiciales. Todas las células tienen unos mecanismos que les permiten detectar las condiciones de su ambiente físico-químico, y responder a ellas activando la expresión de determinados genes e impidiendo la expresión de otros. Esto es lo que se llama la regulación de la expresión génica.
¿Cómo se Investigó?
Esto fue comprobado por Jacob y Monod en los años 50 en experimentos con la especie bacteriana Escherichia coli. Esta bacteria utiliza la molécula de glucosa como "combustible" único a partir del cual obtiene energía (ATP). Si no dispone de glucosa, debe absorber los azúcares que haya asequibles y convertirlos en glucosa, lo que, lógicamente, resulta más lento, complicado y costoso energéticamente.
Si cultivamos E. coli en un medio sin glucosa pero con lactosa [ß-D-galactopiranosil - (1→4) - D-glucopiranosa], la bacteria incorporará la lactosa, la hidrolizará hasta galactosa y glucosa y finalmente convertirá la galactosa en glucosa. Todo esto ha de hacerlo con el concurso de varios enzimas, entre los que están una permeasa, una galactosidasa y una transacetilasa.
Jacob y Monod observaron que si se añadía glucosa al medio de cultivo, estos 3 enzimas prácticamente dejaban de ser sintetizados. Esto era así, porque la bacteria, siguiendo los habituales principios de economía de recursos comunes a todos los seres vivos, incorporaba la glucosa del medio de cultivo y rehusaba utilizar la lactosa.
La conclusión fue que la bacteria debía de disponer de un mecanismo de regulación que hiciese que determinados genes se expresasen o no, según hubiera o no glucosa en el medio de cultivo.
El Operón
La forma concreta en que sucede esta regulación de la expresión génica fue propuesta por Jacob y Monod en 1961 y es la siguiente:
- Los genes se agrupan en operones. Un operón, en sentido 3'→5', que es el sentido en que se lee durante la transcripción, consta de...
- Un gen regulador, que codifica para una proteína denominada represor.
- Una zona promotora, a la que se asocia la ARN-polimerasa.
- Una zona operadora, a la que se asocia el represor.
- Un gen estructural, que es el que codifica para una proteína. (A veces en las bacterias hay varios genes estructurales en el mismo operón: en este caso están uno detrás de otro, se transcriben simultáneamente en un ARNm policistrónico y codifican para proteínas que intervienen en la misma ruta metabólica.)
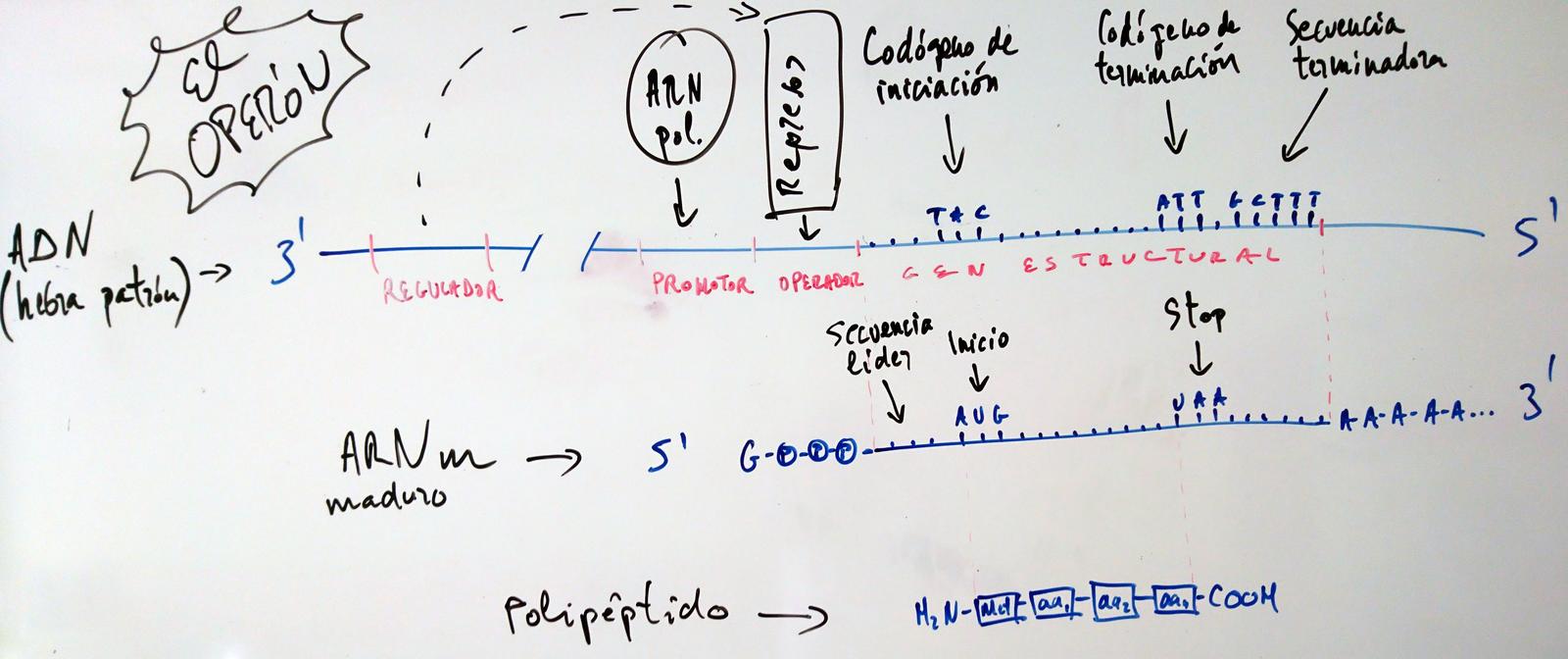
- Cuando un represor está asociado a la correspondiente zona operadora, la ADN-polimerasa no puede avanzar hacia los genes estructurales y por consiguiente estos genes no pueden ser transcritos. El operón en cuestión está, entonces, inactivado.
- Cada operón puede tener uno de los dos siguientes mecanismos de regulación:
- Inducción Enzimática. En este caso la expresión del gen regulador produce un represor activo, que se asocia directamente con la zona operador, inactivando al operón. Pero hay unas moléculas, denominadas inductores, que suelen ser el sustrato de la ruta metabólica catalizada por los enzimas codificados por los genes estructurales del operón, que pueden unirse al represor alterando su conformación, de tal manera que ya no pueda asociarse a la zona operadora, quedando libre el paso a la ADN-polimerasa, que ya puede transcribir los genes estructurales. Este mecanismo de regulación de la expresión génica permite que se sinteticen enzimas solo cuando haya suficiente cantidad del sustrato que han de metabolizar. En el ejemplo del operón de la lactasa, de funcionar de este modo, el inductor sería la lactosa.
- Represión Enzimática = Represión por Producto Final. En este caso la expresión del gen regulador produce un represor inactivo, que no se puede asociar directamente con la zona operador, con lo que la ADN-polimerasa tiene libre el paso hacia los genes estructurales y puede transcribirlos. Pero hay unas moléculas, denominadas correpresores, que suelen ser el producto de la ruta metabólica catalizada por los enzimas codificados por los genes estructurales del operón, que pueden unirse al represor alterando su conformación, de tal manera que ya pueda asociarse a la zona operadora, bloqueando el paso a la ADN-polimerasa, que ya no puede transcribir los genes estructurales. Este mecanismo de regulación de la expresión génica permite que se dejen de sintetizar enzimas cuando estas hayan producido una cantidad suficiente de producto. En el ejemplo del operón de la lactasa, de funcionar de este modo, el correpresor sería la glucosa.
Variaciones en los Eucariontes
En los eucariontes hay mecanismos adicionales de regulación de la expresión génica, entre ellos:
- El grado en que el ADN espaciador de los nucleosomas esté enrollado en torno a la histona H1, haciéndolo más o menos accesible a las ARN-polimerasas;
- La metilación de las bases, que activa o inactiva genes.
Ambos mecanismos son afectados por...
- Los factores epigenéticos, que son condicionantes circunstanciales que inducen la activación o inactivación de genes. Algunos son la edad, la dieta, los fármacos o las sustancias químicas presentes en el ambiente.
- Las hormonas, producidas y liberadas a la sangre por las glándulas endocrinas, tras ser estimuladas por el Sistema Nervioso Central. Las hormonas viajan por la sangre a través de todo el cuerpo, pero solo las células diana de los órganos blanco de cada hormona tienen receptores para cada una (p.e. el hígado es un órgano blanco para la insulina, pero la piel no lo es). Cuando una hormona se asocia a sus receptores en las células diana, desencadena en esta una serie de reacciones metabólicas que tienen como resultado final la activación de uno o de unos determinados operones, produciéndose en el individuo una respuesta fisiológica determinada.
Las Mutaciones
Las mutaciones genéticas son cambios que se producen en el material hereditario de una célula de un individuo, y que traen consigo que la célula mutante vaya a tener un material hereditario diferente del que tenía la célula madre de la que procede. Las células afectadas por una mutación se denominan células mutantes, y los individuos a los que pertenecen individuos mutantes.
En el caso de un individuo pluricelular, como todas las células provienen de una misma célula madre original, todas van a tener en principio el mismo genotipo. Pero si una de ellas sufre una mutación, ella y su descendencia va a tener un genotipo diferente del que tienen todas las demás células del individuo.
Si la célula mutante es una célula somática (como una célula muscular o epitelial, que no va a participar en la reproducción del individuo), entonces la mutación no se va a transmitir a la descendencia del individuo, aunque sí se va a transmitir a las células descendientes de la célula mutante, lo que puede ser causa de un tumor.
Si la mutación se produce en un gameto (como un óvulo o un espermatozoide) que llegue a participar en la producción de un nuevo individuo, entonces la mutación se va a transmitir al individuo hijo y a toda su descendencia. Estas son las mutaciones heredables.
Las mutaciones heredables son la causa de la aparición de nuevos alelos y de nuevos genes, lo que puede dar lugar tanto a la aparición de nuevas clases de individuos de una especie, como a la aparición de nuevas especies. Por ello las mutaciones heredables tienen una enorme importancia en la evolución biológica.
Clasificación Según su Efecto Sobre los Individuos
Progresivas
Incrementan la eficacia reproductora del individuo mutante, esto es, el individuo va a tener mayores facilidades para reproducirse que los demás miembros de la población. Por ello la mutación acabará expandiéndose dentro del acervo genético de la población. Se dice que la selección natural favorece la expansión de dicha mutación. Son la clase menos frecuente de mutaciones.
Neutras
No afectan a la eficacia reproductora del individuo mutante, por lo que la selección natural no va a favorecer ni su expansión ni su eliminación del genoma de la población. Son la clase más frecuente de mutaciones.
Regresivas o Deletéreas
Disminuyen la eficacia reproductora del individuo mutante, esto es, el individuo será estéril o tendrá dificultades para reproducirse. Por ello la mutación acabará desapareciendo o al menos tendrá una presencia muy reducida dentro del acervo de genes de la población.
Letales
Causan la muerte del individuo mutante, el cual no va a dejar descendencia, por lo que la selección natural elimina inmediatamente dicha mutación.
Clasificación Según la Clase de Alteración del Material hereditario
Mutaciones genómicas, cariotípicas o cromosómicas numéricas
Son aquellas que afectan al número de cromosomas de un cariotipo.
Euploidización
La euploidización consiste en una mutación por la que aumenta o disminuye el número de juegos cromosómicos completos de una célula. Hay 2 casos generales: la haploidización, por la que una célula diploide pasa a ser haploide y la poliploidización, por la que aumenta el número de juegos cromosómicos completos de una célula.
La poliploidización, cuando ocurre en células germinales, es causa de aparición inmediata de una nueva especie (especiación cuántica, que es lo opuesto a especiación gradual) que puede o no prosperar. De hecho es una forma muy frecuente de especiación entre las plantas, aunque es muy rara entre los animales. Las plantas poliploides suelen ser más grandes, resistentes y capaces de colonizar nuevos hábitats. Por ejemplo, el origen del trigo común o harinero (Triticum aestivum), que es hexaploide (6 juegos de 7 cromosomas), está en dos eventos consecutivos de poliploidización en sus especies ancestrales.
Aneuploidización
Es la mutación que causa que haya algún cromosoma de más o de menos respecto al cariotipo normal. Se utilizan los términos nulisomía, trisomía, tetrasomía, etc., cuando en lugar de los dos cromosomas de un par homólogo no hay ninguno o hay tres, cuatro, etc., respectivamente. En la especie humana son características las siguientes:
- Síndrome de Down. Es una trisomía del cromosoma XXI. Presentan deficiencias físicas y mentales, y los varones son estériles.
- Síndrome de Klinefelter. En vez de un par sexual normal (X-X o X-Y) presentan la dotación X-X-Y. Varones estériles con escaso desarrollo genital y retraso mental.
- Síndrome de Turner. La dotación de cromosomas sexuales se reduce a un único cromosoma X. Mujeres estériles con escaso desarrollo genital y retraso en el crecimiento.
- Síndrome del duplo Y. En vez de un par sexual normal presentan la dotación X-Y-Y. Varones corpulentos, con retraso mental y con tendencia a ser violentos.
Fusión cromosómica
Es la fusión de dos cromosomas en uno solo. Ha sido un fenómeno muy frecuente en la evolución de los primates (el chimpancé tiene 24x2 cromosomas y nosotros 23x2, por lo que cabe suponer que algún ancestro nuestro sufrió una fusión cromosómica, y que esa mutación contribuyó a la aparición de nuestra especie).
Fisión cromosómica
Es la división de un cromosoma en dos fragmentos, uno de los cuales conservará el centrómero del cromosoma original. En el otro fragmento, para no perderse, ha de aparecer un nuevo centrómero.
Mutaciones cromosómicas
Son aquellas que afectan a la estructura de un cromosoma.
Se pueden detectar al microscopio óptico durante la profase I de la meiosis, que es cuando se aparean los cromosomas homólogos, ya que en los bivalentes van a aparecer regiones desapareadas o con apareamientos anómalos, que dan lugar a bucles o formaciones en cruz.
Delección
Es la pérdida de algún segmento de algún cromosoma, y con ello, de un fragmento de una molécula de ADN que puede contener información genética importante, por lo que normalmente son regresivas o letales.
En la profase I de la meiosis el cromosoma completo va a tener una región desapareada a la altura de la delección de su homólogo (se forma un bucle o hay un extremo desapareado).
Duplicación
Es la repetición de un mismo segmento cromosómico un determinado número de veces, dentro de un mismo cromosoma. Los segmentos repetidos pueden estar situados en tándem (consecutivos) o separados.
En la profase I de la meiosis el cromosoma con el segmento duplicado va a tener una región desapareada a la altura de la duplicación (se forma un bucle o hay un extremo desapareado).
Cuando han afectado al ADN silencioso han dado origen a las secuencias de ADN repetitivo que se encuentran, principalmente, en los telómeros y en el centrómero.
Y cuando afectan a genes difícilmente son letales o regresivas. De hecho, su importancia en la evolución biológica es enorme. Esto es así porque una duplicación puede afectar a un gen completo, con lo que ahora tendremos dos copias idénticas del mismo. Tras ello la selección natural podría favorecer...
- la estabilidad de las dos copias idénticas del locus duplicado, para que así se pueda sintetizar más cantidad de la misma proteína o ARN; es el caso de los genes que codifican para los ARNt o para el ARNr, que se encuentran repetidos varias docenas de veces;
- la evolución divergente de una de las dos copias, de modo que pase a codificar para una proteína que determine un carácter fenotípico nuevo. En este caso habrá aparecido un nuevo gen.
Inversión
Un segmento de un cromosoma gira 180º respecto a su posición inicial tras escindirse y reasociarse al cromosoma en el mismo punto, pero en posición invertida. Si el segmento invertido incluye al centrómero, se llama inversión pericéntrica; si no lo incluye, se llama inversión paracéntrica. En la profase I de la meiosis van a aparecer bucles. Como van a favorecer sobrecruzamientos aberrantes, pueden producir individuos anormales.
Translocación
Consiste en la escisión de un fragmento de un cromosoma que se desplaza a una posición nueva en el mismo o en otro cromosoma. Si hay un intercambio de segmentos cromosómicos, que es lo más frecuente cuando la translocación ocurre entre dos cromosomas diferentes, se llama translocación recíproca. Si solo se desplaza un único fragmento se llama transposición. En la profase I de la meiosis van a aparecer formaciones en cruz. Como van a favorecer sobrecruzamientos aberrantes, pueden producir individuos anormales.
Mutaciones génicas o puntuales
Son aquellas que afectan a uno o más nucleótidos o bases de un gen.
Sustituciones de unas bases por otras
- Si suceden en los intrones de un gen, carecerán de efectos fenotípicos.
- Si suceden en los exones de un gen producirán el cambio de un codógeno por otro. En este caso...
- Pueden dar lugar a la sustitución de un codógeno por otro que codifique para el mismo aminoácido, en cuyo caso la mutación carecerá de efectos fenotípicos.
- Pueden dar lugar a la sustitución de un codógeno por otro que codifique para un aminoácido diferente, en cuyo caso la función biológica de la proteína se verá afectada si el aminoácido sustituido pertenecía al centro activo de una proteína enzimática o era esencial para el mantenimiento de la conformación.
- Pueden dar lugar a las llamadas "mutaciones sin sentido":
- Si la sustitución altera el codón de iniciación, el polipéptido dejara de sintetizarse.
- Si la sustitución altera el codón de terminación, el polipéptido tendrá aminoácidos de más y su función se verá totalmente alterada.
- Si la sustitución genera un codón de terminación, el polipéptido tendrá aminoácidos de menos y su función se verá totalmente alterada.
Delecciones, inserciones y duplicaciones de nucleótidos
Si afectan a 3 nucleótidos a un número de ellos múltiplo de 3, causarán la pérdida o ganancia de algún aminoácido, con lo que la proteína resultante perderá su función biológica original.
Si afectan a un número de nucleótidos distinto de 3 o de múltiplo de 3, causarán el corrimiento en el orden de lectura de las bases durante la traducción; es decir, a partir del punto en que se produjo la mutación, los codones (y los aminoácidos) serán diferentes de los originales. La proteína resultante también perderá su función biológica original.
Consecuencias de las mutaciones génicas
Como veremos más adelante, existe un eficaz mecanismo de reparación de las mutaciones que afectan solo a uno o unos pocos nucleótidos, lo que reduce en unas 10.000 veces su frecuencia. De aquellas que no llegan a ser corregidas, la mayoría son silenciosas: carecen de efectos fenotípicos. Esto es así porque...
- la mayoría (un 90%) del ADN de un genoma eucariótico es silencioso (su secuencia es irrelevante de cara al funcionamiento de las células): pseudogenes, transposones, ADN repetitivo...;
- los genes no codificantes (los que dan lugar a los ARNt y los ARNr) están muy repetidos; y
- los genes codificantes eucarióticos incluyen regiones no codificantes: los intrones.
Por consiguiente, para que una mutación génica no sea silenciosa, habrá de afectar a un elemento regulador o a un exón de un gen que codifique para una proteína. Y además la mutación habrá de alterar la proteína de tal forma que afecte a su función biológica, impidiéndola, dificultándola o mejorándola, en cuyo caso podremos decir que se ha generado un nuevo alelo. Finalmente, su impacto fenotípico dependerá de que...
- el gen mutado esté poco o nada repetido;
- la proteína codificada por el gen mutado tenga un papel importante para la vida del individuo.
Clasificación Según su Causa
Espontáneas
Son debidas a...
- Errores durante la replicación del ADN: las polimerasas de ADN colocan una base errónea de cada 10.000. Si el error no es corregido, es causa de una sustitución.
- El comportamiento anómalo de los cromosomas durante la meiosis o durante la mitosis puede ser causa de todo tipo de mutaciones cromosómicas o cariotípicas.
- Despurinaciones: son pérdidas de bases púricas (A o G) al destruirse, por acción del calor, el enlace N-glucosídico entre la base púrica y la desoxirribosa. Queda un hueco, y cuando haya una replicación o una transcripción de ese sector de ADN, la polimerasa (de ADN o de ARN) no va a saber que base colocar en el polinucleótido que está sintetizando al llegar a este punto, y colocará una cualquiera al azar, lo que es causa probable de una sustitución.
- Desaminaciones: son pérdidas espontáneas de grupos amino de una determinada base, que se transforma en otra diferente, la cual puede aparearse con una base distinta, lo que nos produce una sustitución. Así por ejemplo la C (que se aparea con la G) al desaminarse se transforma en U (que se aparea con la A).
Inducidas
Causadas por agentes mutágenos, muchos de los cuales se encuentran en exceso en el medio ambiente urbano a causa del desarrollo tecnológico. Dichos agentes pueden ser:
- Las radiaciones ionizantes, como los rayos X, partículas α, partículas ß y rayos γ.
- La radiación ultravioleta, que produce la asociación entre dos bases pirimidínicas contiguas en la misma hebra de ADN, como los dímeros de timina. Dichas bases ya no se aparean por enlaces de puente de hidrógeno con las bases complementarias que tienen enfrente. Y cuando haya una replicación o una transcripción de ese sector de ADN, la polimerasa (de ADN o de ARN) no va a saber que base colocar en el polinucleótido que está sintetizando al llegar a este punto, y colocará una cualquiera al azar, lo que es causa probable de una sustitución.
- Los ultrasonidos.
- Agentes químicos, como el HNO2, el gas mostaza, la nicotina, y diversos fármacos y pesticidas.
- Virus (retroviris, hepatitis B...) y transposones (segmentos saltadores de ADN). Al insertarse dentro de genes reguladores o codificantes pueden activar o inactivar a estos últimos.
La Reparación de Errores en el ADN (No EBAU)
En genomas tan grandes como el humano (6,4 x 109 pb en una célula diploide femenina) la probabilidad de que haya una mutación génica en alguno u otro lugar de nuestro ADN es muy alta, ya sea una mutación espontánea (por errores durante la replicación) o inducida (por algún agente mutágeno, como la luz ultravioleta). De hecho este tipo de mutaciones se está produciendo continuamente en nuestras células. Por ello, de no haber una eficaz serie de mecanismos de reparación de los errores en la secuencia de bases del ADN, la consecuencia sería una inestabilidad tal de la información genética de la especie, que probablemente esta no podría subsistir. Son los siguientes:
- En primer lugar las ADN-polimerasas son capaces de reconocer y corregir sus propios errores, mientras están replicando el ADN, en un 99%: si han colocado un nucleótido equivocado, su base no quedará apareada con la de la hebra patrón situada enfrente; la ADN polimerasa "percibe" este desapareamiento de la base situada "a sus espaldas" y, gracias a su actividad exonucleasa, inmediatamente la sustituye por la correcta.
- En segundo lugar cuando una molécula de ADN tiene una porción alterada (p.e. sin bases, tras una despurinación; o sin apareamientos entre bases enfrentadas, tras formarse un dímero de timina o tras una desaminación), se activa el siguiente mecanismo de reparación: una enzima endonucleasa realiza una muesca al lado de la zona alterada (es decir, corta un enlace fosfodiéster). Acto seguido, una ADN-polimerasa, gracias a su actividad exonucleasa, elimina los nucleótidos de dicha zona uno a uno. Después, la misma enzima, con su actividad polimerasa, coloca los nucleótidos correctos. Y por último una enzima ligasa sella los huecos entre nucleótidos contiguos de la hebra reparada (fabrica el correspondiente enlace fosfodiéster).
- En tercer lugar hay unas enzimas especiales, las ADN-glicosilasas, que reconocen y eliminan las bases alteradas al hidrolizar el enlace N-glucosídico que une a la base con la desoxirribosa. Tras ello queda un hueco en la hebra de ADN afectada, que terminará de ser reparado por el mecanismo explicado en el párrafo anterior. Cada ADN-glucosilasa reconoce específicamente a un tipo concreto de base alterada (timina, guanina...).
Esto acaba dando lugar a que, tras la replicación del ADN, la proporción de errores sea de tan solo 1 por cada 1010 nucleótidos.
Algunos Avances Decisivos en la Historia de la Genética (No EBAU)
- Mendel demuestra en 1865 la existencia de los genes, sin que su trabajo tenga trascendencia alguna.
- En 1900 De Vries, Correns y Tschermack redescubren, trabajando independientemente, las reglas de la herencia propuestas por Mendel.
- Morgan demostró en 1905 que los genes se encuentran alineados en lugares concretos de los cromosomas.
- Avery, MacLeod y McCarty demostraron en 1944 que el ADN de los cromosomas era el portador de la información genética, tras estudiar las transformaciones bacterianas que fueron observadas por Griffith en 1928.
- Beadle y Tatum propusieron en 1948 el axioma fundamental que resumía el funcionamiento de la información genética: "un gen - un enzima". Se le llamó el "dogma central de la Biología Molecular".
- Watson y Crick propusieron en 1953 el modelo de la doble hélice para la estructura secundaria de la molécula de ADN, apoyándose en los estudios de las placas de difracción de rayos X del ADN hechos por Franklin y Wilkins.
- Arthur Kornberg aisló y descubrió en 1956 a la enzima ADN-polimerasa como responsables de la replicación del ADN.
- Meselson y Stahl comprobaron en 1958 que la replicación del ADN seguía el modelo semiconservativo, que había sido sugerido previamente por Watson y Crick.
- Samuel Weiss y Jerard Hurwits aislaron e identificaron en 1960 a la enzima ARN-polimerasa como responsable de la transcripción del ADN.
- Jacob y Monod proponen en 1961 el mecanismo de regulación de la expresión génica basado en el operón.
- Niremberg, Ochoa y otros, entre 1961 y 1965, dedujeron la serie de correspondencias entre tripletes de nucleótidos y aminoácidos que constituye el código genético.
- Cairns, en 1963, observó las horquillas de replicación del ADN en placas de autorradiografiado.
- Okazaki propuso en 1968 el modelo de replicación fragmentada de la hebra retardada de cada horquilla de replicación.
- Arber, Nathans y Smith, a finales de la década de 1960, descubren las endonucleasas de restricción de las bacterias y arqueas, lo que condujo, junto con el descubrimiento de las ligasas, al desarrollo de la tecnología de ADN recombinante o Ingeniería Genética. El primer uso práctico de su trabajo fue la manipulación de la bacteria E. coli para producir insulina humana para los diabéticos.
- Francis Mojica descubre en 1993 las secuencias CRISPR del genoma de las arqueas y las bacterias, cuyo conocimiento permitió el desarrollo del método de edición genética basado en las enzimas CRISPR-Cas 9, ideado por Emmanuelle Charpentier y Jennifer Doudna en 2012.